# 介绍
揭示使用 PyMOL 这一行业领先的分子可视化软件制作令人惊叹的蛋白质图形的秘密。在这篇中级水平的博客中,我们将深入探索一系列无比宝贵的技巧和窍门,助您将科研可视化提升到全新高度。了解如何利用 PyMOL 的高级功能创建惊艳、适合发表的高质量图像,增强结构清晰度,并通过精美的三维模型吸引观众的注意力。无论您是经验丰富的研究者还是崭露头角的科学家,这篇博客都是您掌握 PyMOL 并将蛋白质图形转化为视觉杰作的必备指南。
蛋白质可视化 PyMol 提供了多种不同的方式来可视化我们的蛋白质和分子。我们将要介绍的重要方法包括卡通表示、主链表示、主链+侧链表示以及表面表示。
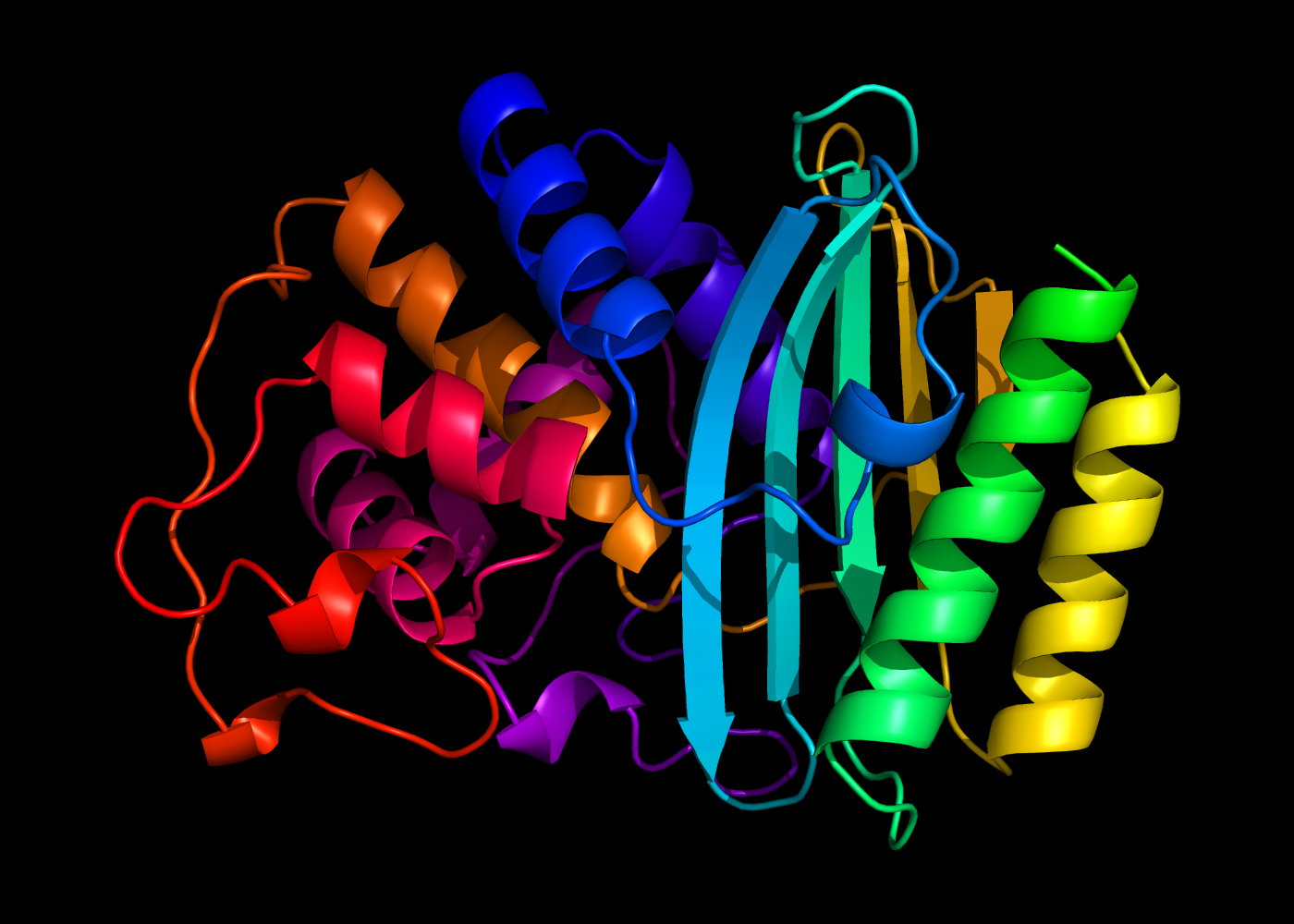
# 卡通表示:
最常用的蛋白质和复合物三维表现形式可能是卡通模型。卡通可视化经常被使用,因为它提供了直观且全面的蛋白质结构视图。此外,卡通模型是展示蛋白质二级结构(如 β-片层和 α-螺旋)的直观方式,也是 PyMol 的默认可视化选项。
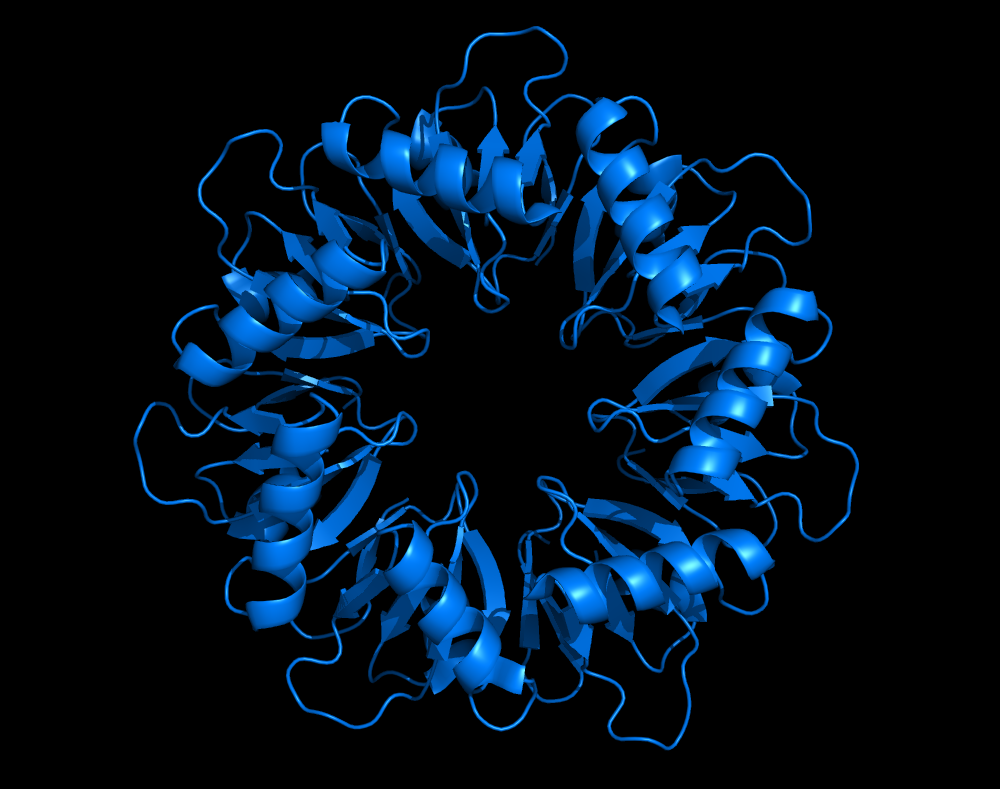
7BME蛋白的卡通模型
# 主链表示:
仅主链表示是一种极简主义的表现形式,只包含了蛋白质主链内部的原子。

7BME蛋白的主链模型(要显示主链,请点击蛋白质对象上的显示选项卡 - >主链 - >条或线)
# 主链+侧链表示:
主链+侧链可视化则是一个非常详细的视图,能够有效地显示蛋白质内部的所有原子。
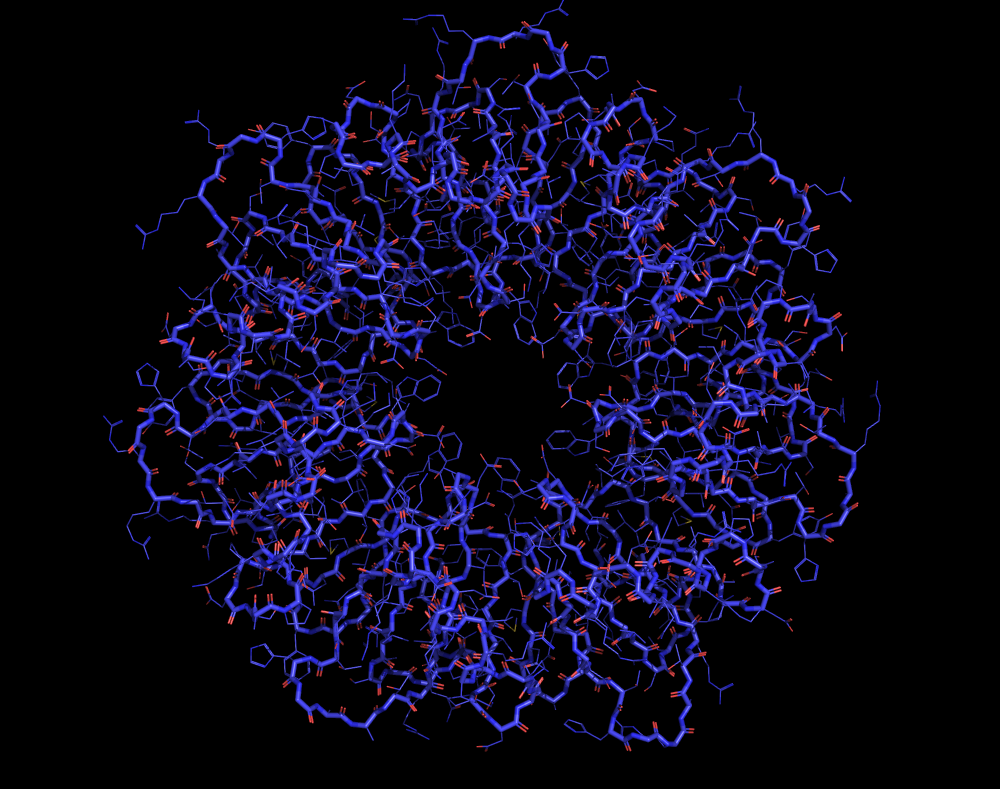
7BME蛋白的主链 + 侧链模型(要显示侧链,请点击蛋白质对象上的显示选项卡 - >侧链 - >条或线)
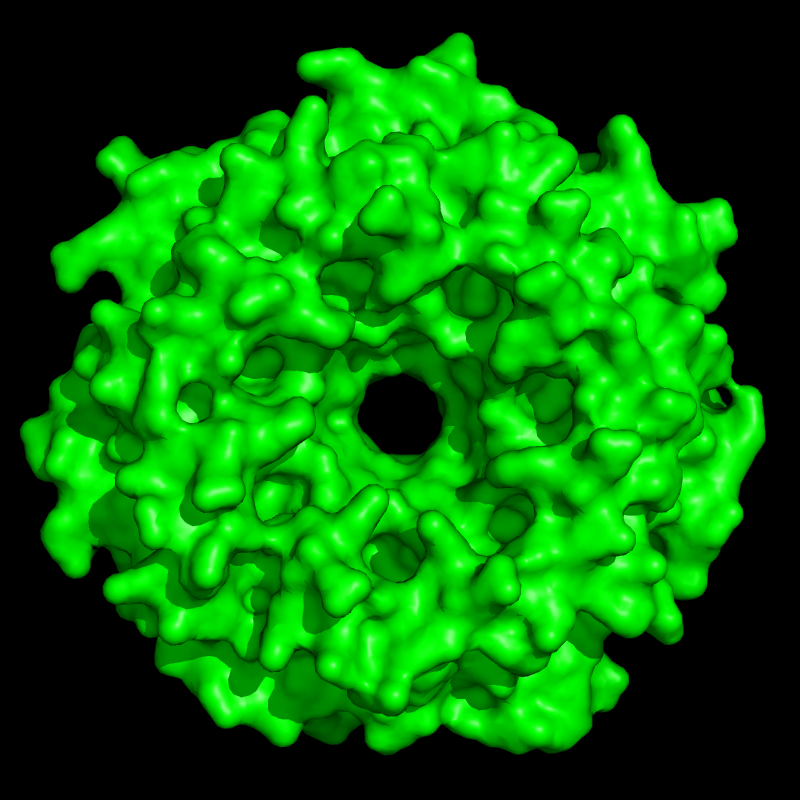
# 表面表示:
表面可视化是指在所有可能位置与蛋白质接触的水分子所构成的表面。
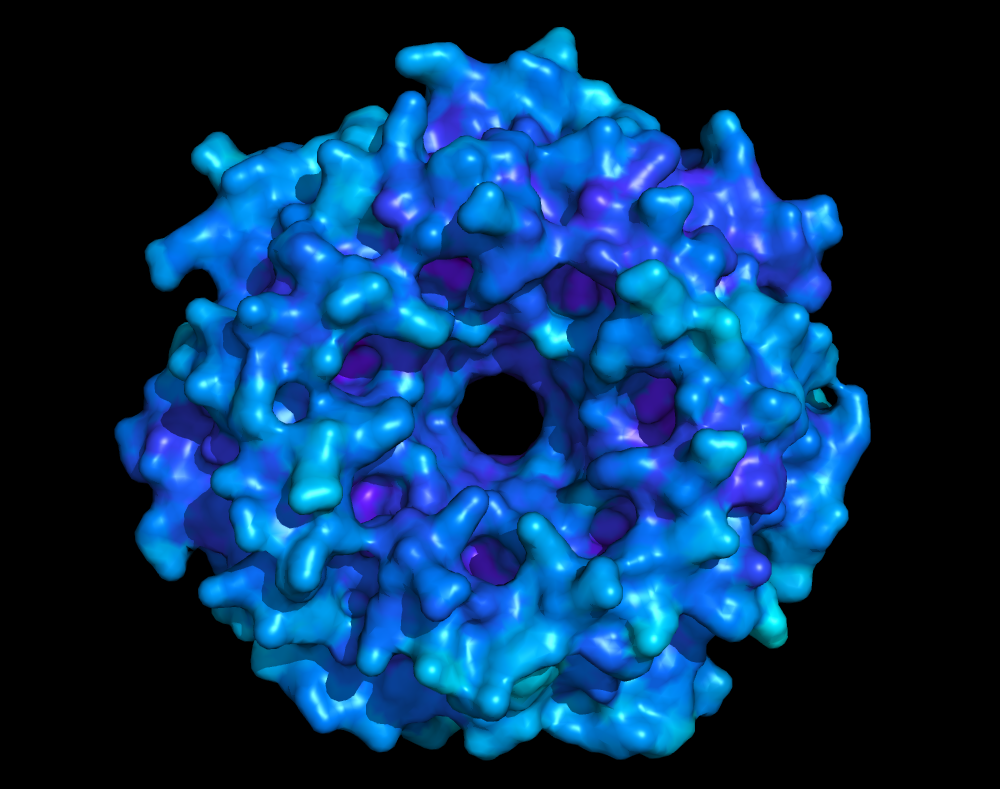
7BME蛋白的表面模型
关于表面显示,另一值得关注的是,您可以轻松设置其透明度并与卡通或主链等其他可视化方式结合使用。设置透明度的命令示例:set transparency, [opacity], [protein_object_name],例如 set transparency, 0.75, 5tvn。
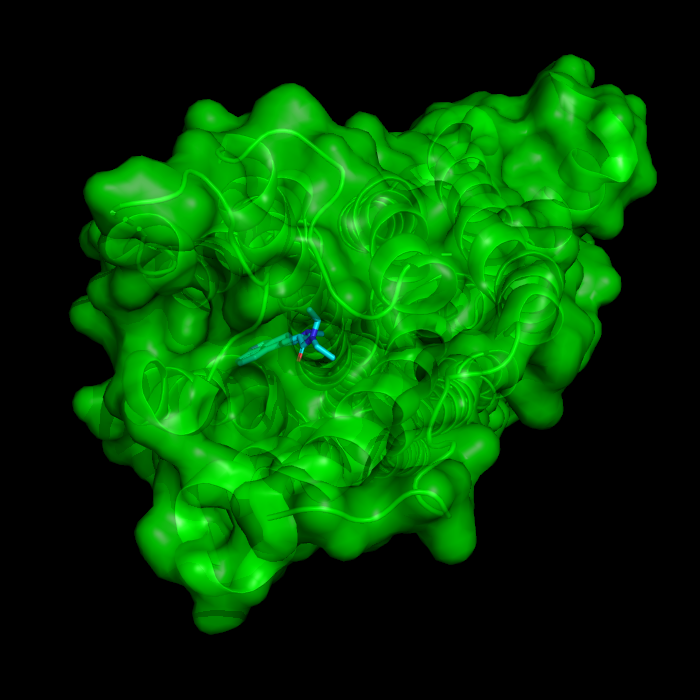
LSD与5-HT2b人血清素受体(5TVN)结合(蛋白质受体的不透明度设置为0.75,使下面的卡通表示也可见)
# 按 B 因子字段着色
pdb 和 mmcif 文件中的 B 因子字段(有时也称为温度因子字段)可视为对每个残基额外的度量指标或元数据,在许多情况下能为我们提供关于给定结构潜在不准确性的有用信息。对于实验结构,它反映了大分子结构中原子的运动性和灵活性,体现了这些原子相关的热振动特性。
传统上,B 因子代表了蛋白质或其他大分子结构中每个原子的温度因子或热振动因子。B 因子提供了有关结构中原子移动性或柔韧性信息。B 因子值衡量由于热波动导致原子从平均位置偏离的程度,它指示了原子位置的不确定性或“模糊”。较高的 B 因子值通常对应较高的原子活动性或柔性,而较低的 B 因子值则表明原子位置更为受限或明确。B 因子通常以平方埃(Å^2)为单位,并可通过 X 射线晶体学或核磁共振(NMR)光谱学等技术实验证明得到。
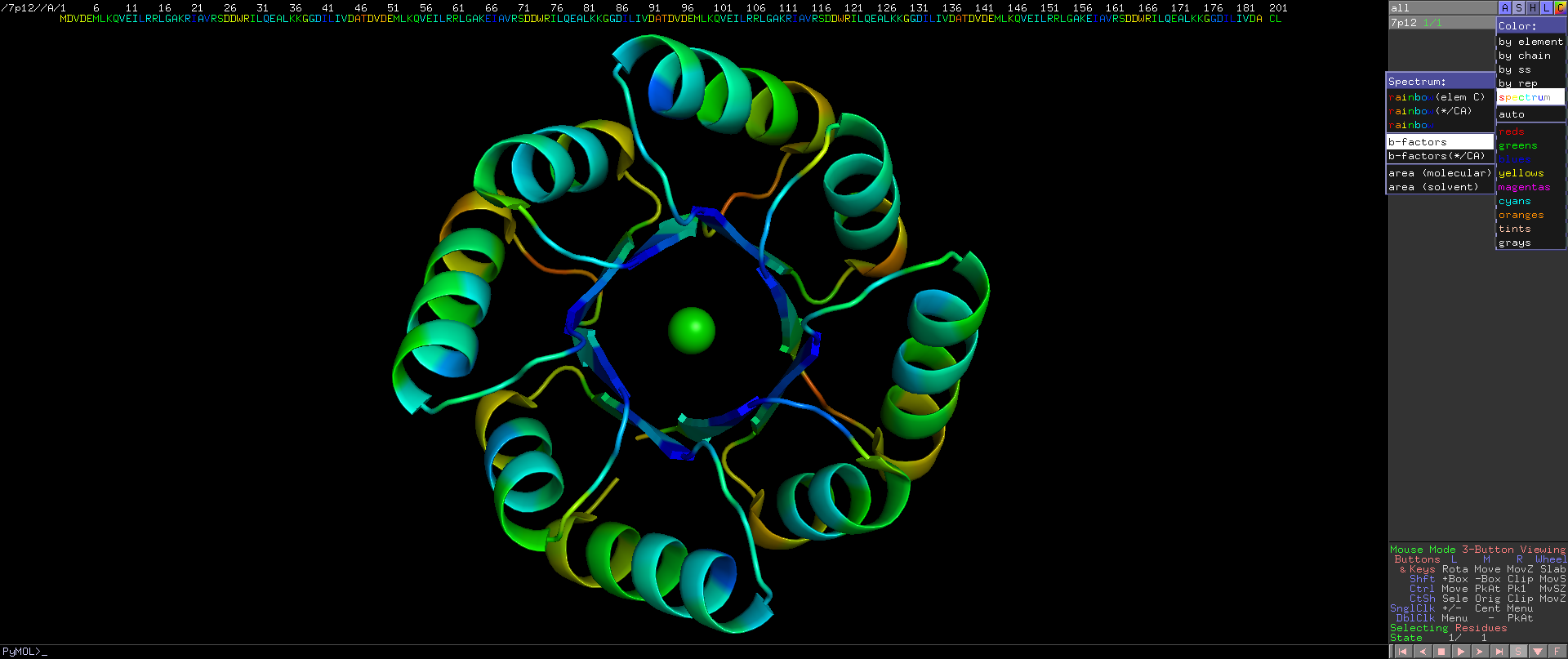
用盐桥簇 (7P12) 为重新设计的 TIM(桶着色要给蛋白质物体着色,只需点击颜色选项卡 - >光谱 - > b 因子)
# 基于 AlphaFold2 预测的 pLDDT 着色
AlphaFold2 预测的结构也有 B 因子字段,但这些值对应于每个残基的 pLDDT 值,其中越高越好。要在 PyMol 中根据 pLDDT 值对 AlphaFold2 结构进行着色,请输入以下命令:Command: spectrum b, tv_red_yellow_green_cyan_marine, minimum=50, maximum=90。
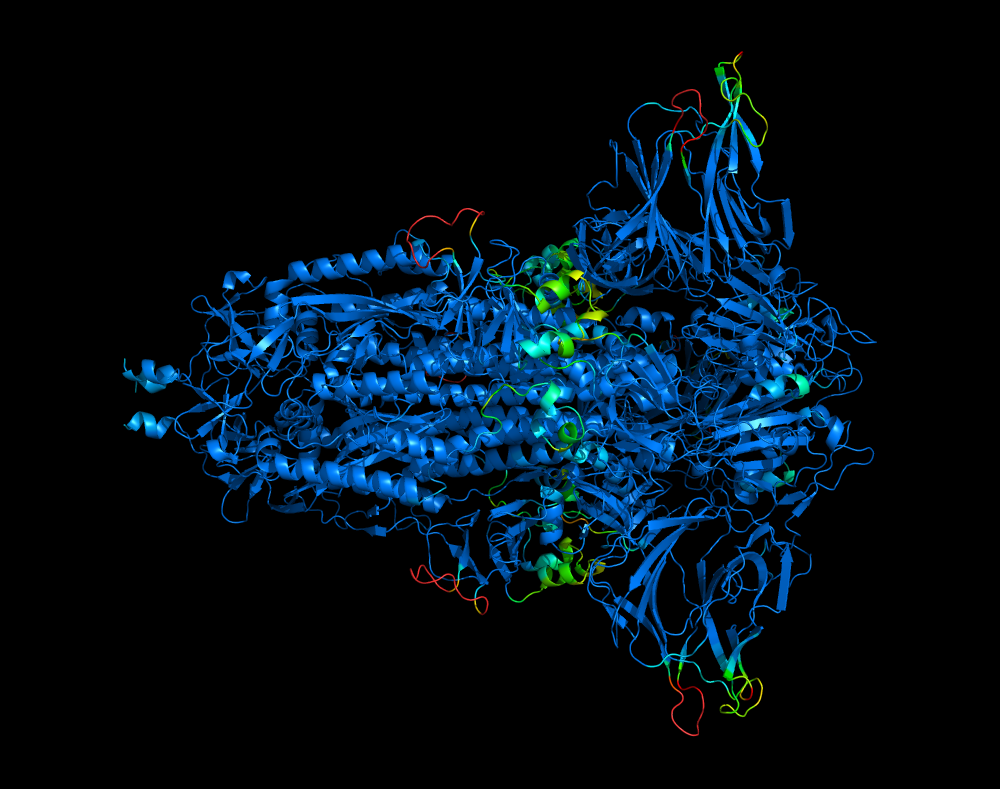
AlphaFold2 预测刺突糖蛋白的结构,由 pLDDT 场着色
# 基于 RoseTTAFold 预测的 pLDDT 着色
同样,RoseTTAFold 也使用自身的 pLDDT 版本填充 B 因子字段,对其结构进行着色的命令如下:Command: spectrum b, tv_red_yellow_green_cyan_marine, minimum=0.5, maximum=0.9。
# 更多频谱选项
此外,其他一些很酷的光谱选项包括按链着色、按原子数着色等,有关更多完整列表选项请访问PyMol wiki (opens new window)。
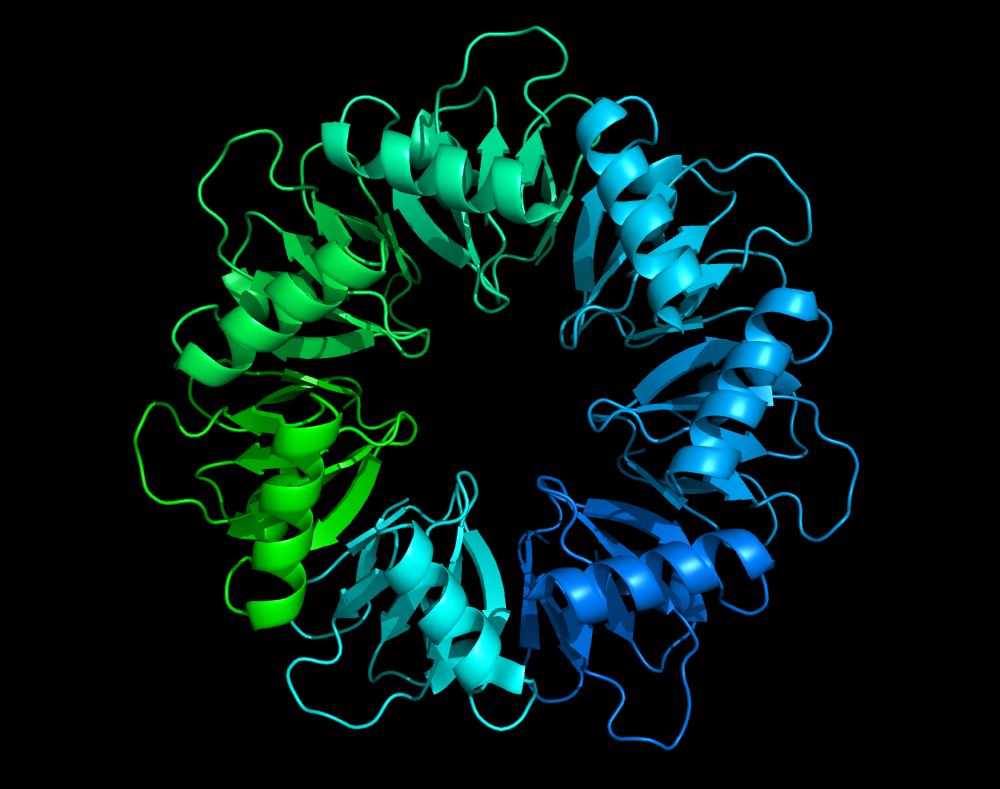
7BME使用命令通过链着色的示例 在这种情况下,链的颜色是使用命令中定义的颜色进行插值的(spectrum chain, green_cyan_marine)
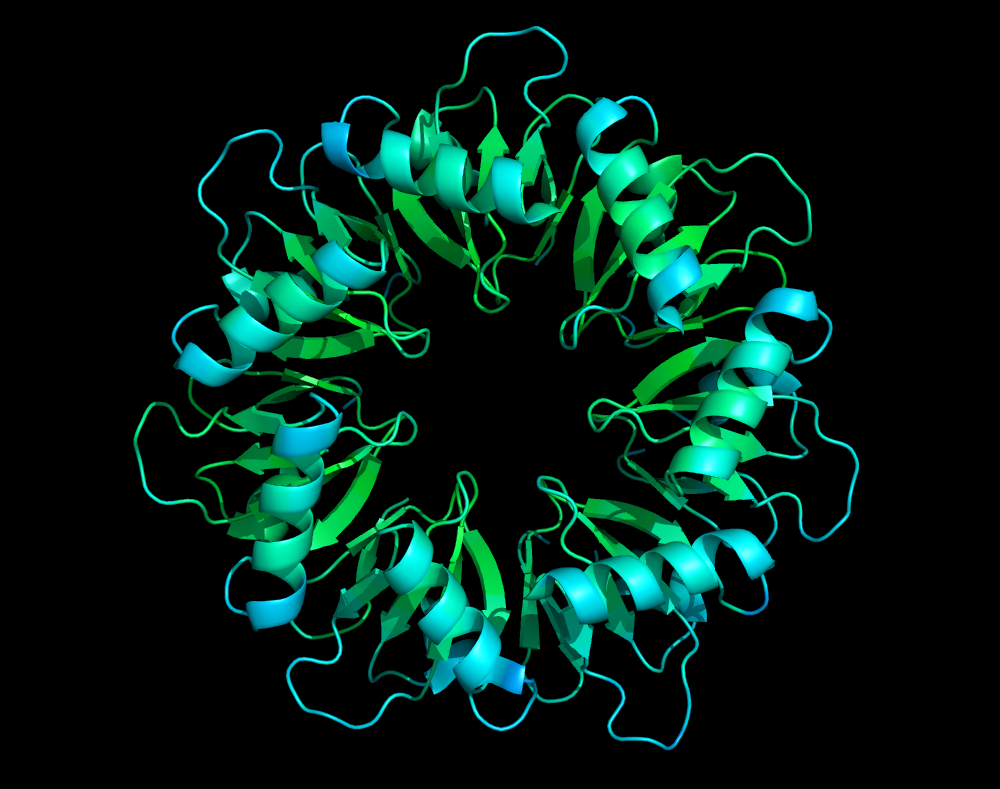
使用命令按B因子对7BME着色的示例 在这种情况下,残基的颜色是使用B因子字段中定义的颜色进行插值的,从而产生引人注目的视觉效果(spectrum b, green_cyan_marine)
这些着色选项也可以应用于蛋白质表面,可以通过转到显示选项卡 –> 表面来切换
使用非常漂亮的梯度对蛋白质的主链进行着色(spectrum resv, yellow_orange_red_magenta_blue_cyan_green)
# 对药物和配体建模
对于一个复合物或多聚体,实际上可以显示距离配体分子 5 埃范围内的所有侧链。命令示例:show sticks, byres all within 5 of [ligand_object],例如 show sticks, byres all within 5 of ligand。
我们选择 5 埃作为截至值,因为氢键是从一个原子的中心到另一个原子的中心(原子核到原子核)测量的。如果以埃为单位测量,氢键的距离通常为 2.8 到 3.4 埃。离子相互作用可以稍短一些,非极性链之间的伦敦力或范德霍尔相互作用通常在 3.8 到 4.2 范围内。这就是上述 选择 5 埃的原因。
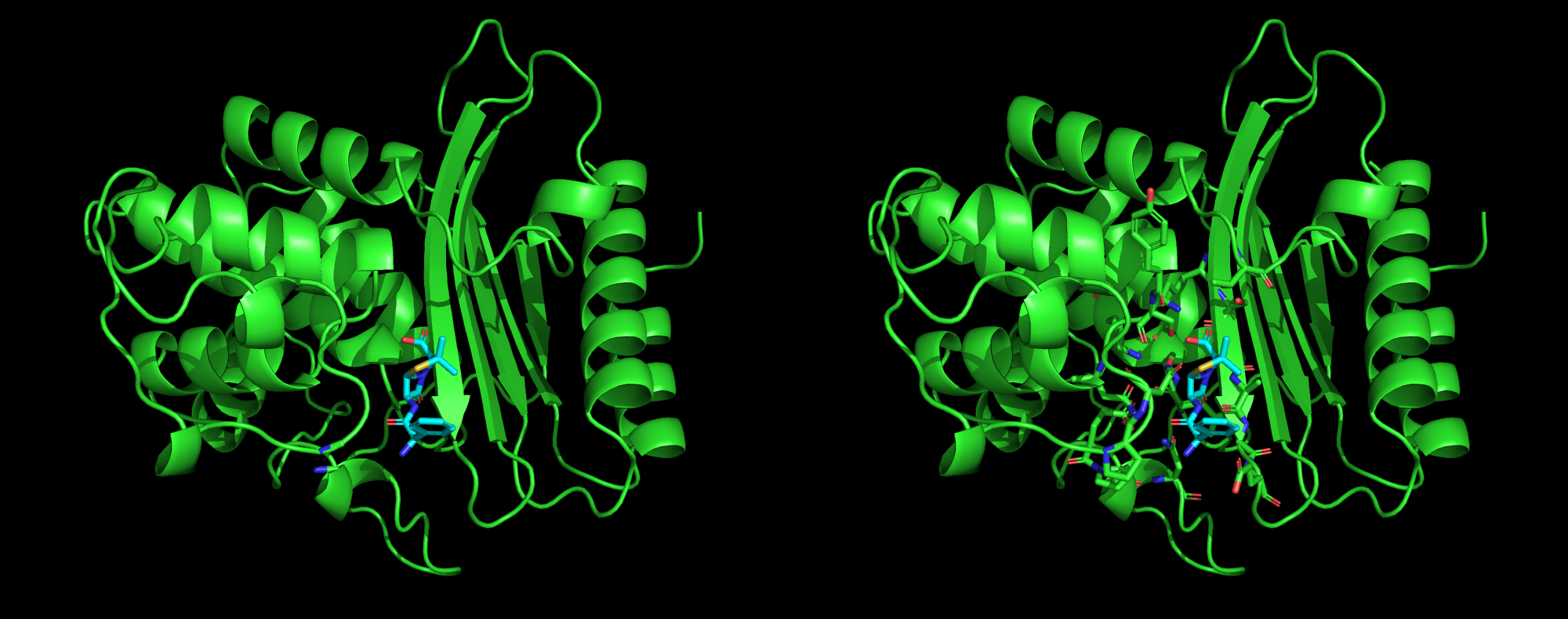
以卡通表示渲染的复杂特写与同时显示侧链比较
另一个非常有用的技术是显示所有极性相互作用/氢键。
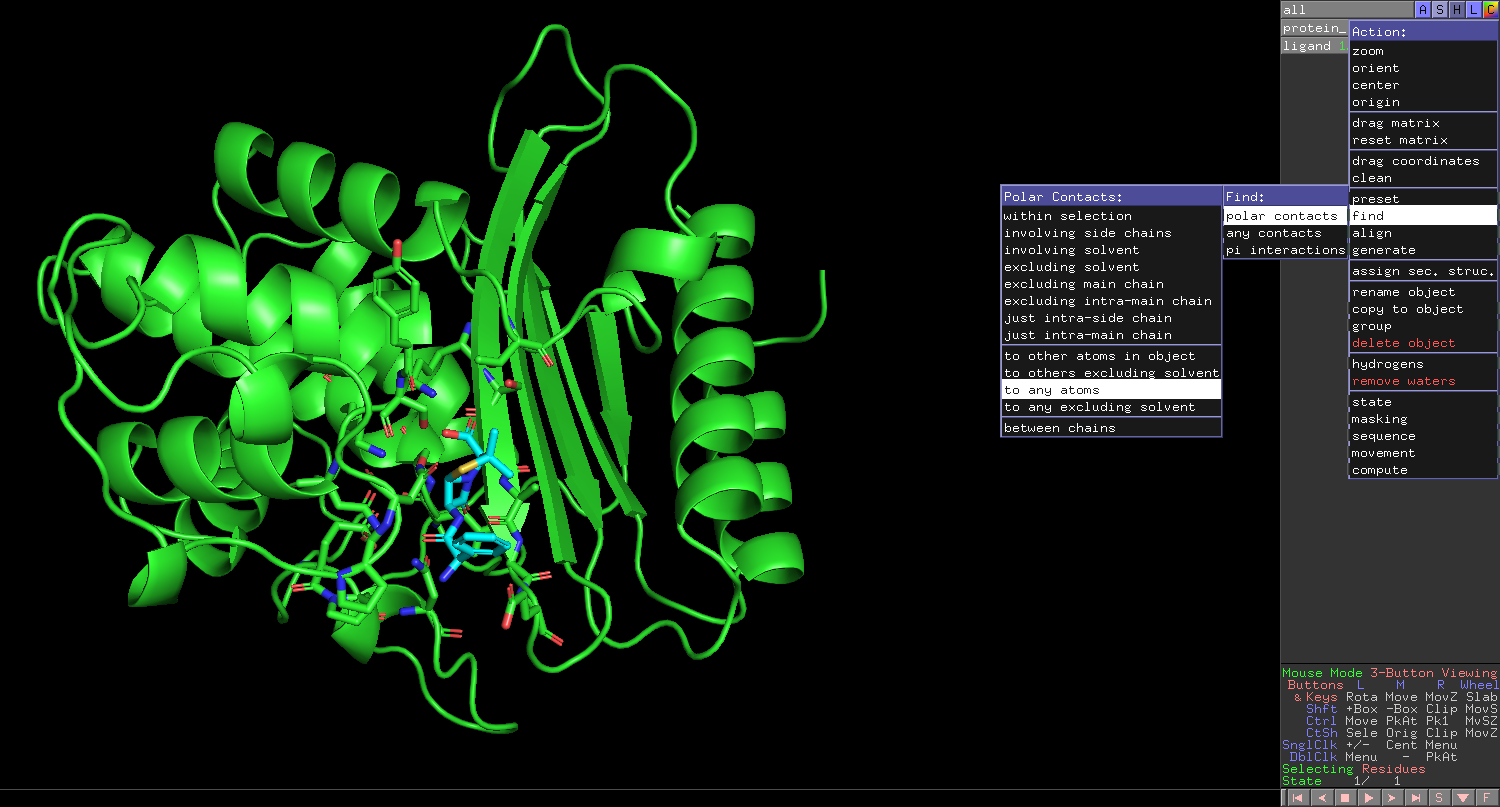
显示氢键的说明(点击受体蛋白的动作按钮,然后导航以查找 ->极性接触 - 与任何原子->用氢键呈现为黄色虚线的复合物(左图)渲染的复杂特写(右图)。)
# 结论
总之,PyMOL 被证明是科研人员用来创建引人注目且信息丰富的蛋白质图形不可或缺的工具。通过运用本博客中探讨的技巧和窍门,您可以解锁 PyMOL 的全部潜能,将科研可视化提升至全新的层次。从操控分子结构到应用先进的渲染技术,现在您已经掌握了制作精美、符合发表标准的图像的知识,这些图像能有效传达您的研究成果。
因此,大胆拥抱 PyMOL 的力量,尝试其丰富多样的功能,并让你的蛋白质图形讲述引人入胜的故事,吸引观众并推动科学可视化的边界。通过实践、专注以及这些专家见解,您正朝着成为 PyMOL 高手的道路迈进,在分子生物学领域留下持久的影响。

